国家微生物组数据协作组织NMDC调研
1、前言
因最近的项目需要涉及微生物组领域的大数据管理,于是在调研这方面的内容,发现美国在2019年开始了这方面的工作,发起了NMDC(https://data.microbiomedata.org)项目,随即中国也不甘落后,中科院微生物所建设了https://nmdc.cn/,后来得知是委托“极道科技”建设的。我对NMDC的总体技术方案做了一些概览性调研。
2、美国NMDC
什么是NMDC?
国家微生物组数据协作项目(NMDC)是2019年6月在美国启动的一项新的试点项目,旨在通过协作的综合数据科学生态系统支持微生物组数据的探索和发现。主要目标是通过提供对多组学微生物组数据的访问来实现微生物组数据科学的民主化,以支持符合FAIR数据原则的可重复的交叉研究分析。
什么是FAIR?
可查找、可访问、可互操作和可重用;
Findability, Accessibility, Interoperability, and Reusability;
NMDC项目的精髓是什么?
利用唯一标识符,将同一个样本分散在不同数据库中的数据连接起来,在NMDC User Portal中可以方便地检索这些关联的数据,给科研带来效率提升。
比如,在JGI GOLD(Genomes OnLine Database)数据库和IMG/M(Integrated Microbial Genomes and Microbiomes)数据库中维护着宏基因组和宏转录组数据,EMSL的MyEMSL/NEXUS系统中维护着宏蛋白质组、代谢组和天然有机表征数据,那么同一个样本的数据就分散在这些不同的数据库里,如果要对一系列样本进行全面和深入的研究,就需要人肉查询各种数据库,NMDC的宗旨就是解决这个问题。
2.1、Document
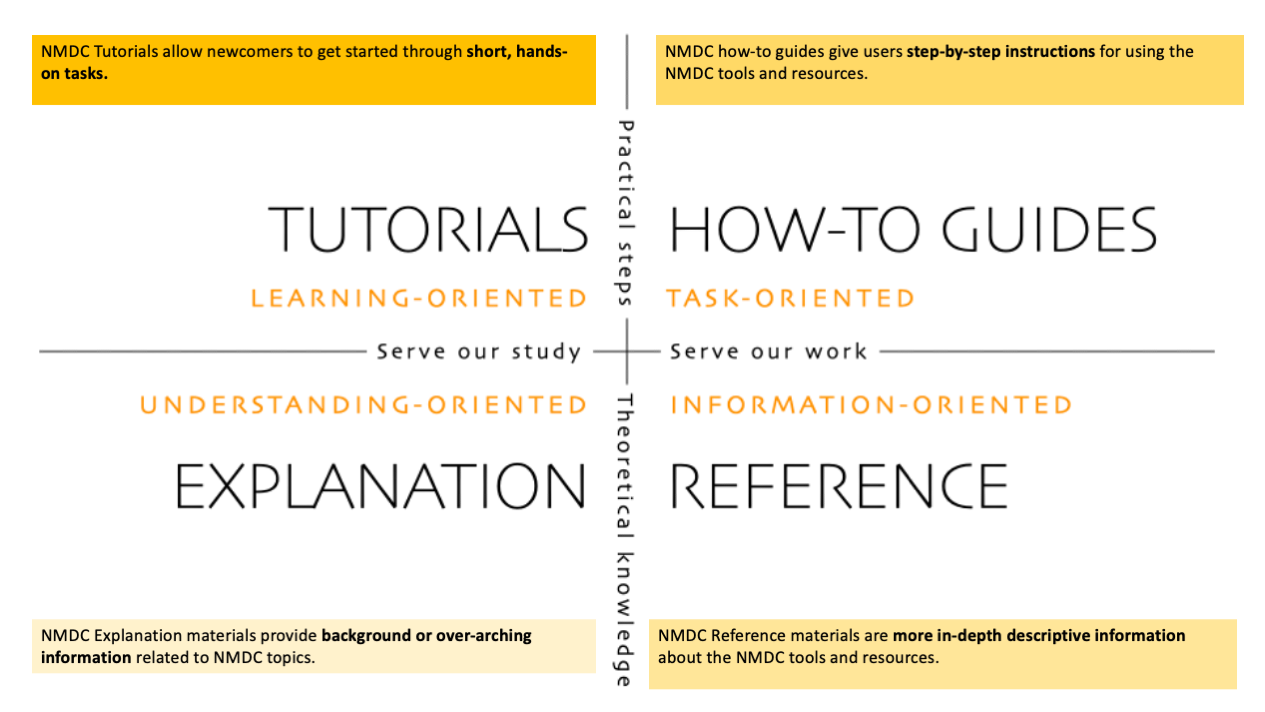
NMDC的文档写的还是很有体系的,第一次见到这么解释自己的文档体系的,毕竟是做学术的。
2.2、MetaData
提交样本信息必须带上MetaData,这是实现样本数据完整性和实现各数据库中的数据交叉关联的基本要求。
Introduction to Metadata and Ontologies- National Microbiome Data Collaborative 这里有关于MetaData的概念解释和重要性解释。
2.3、Schema
提交MetaData必然需要一定的规范,而这个规范就叫Schema(以LinkML YAML格式描述),MIxS就是这种具体的Schema。microbiomedata/nmdc-schema: This repository contains the LinkML specification for the NMDC schema and the artifacts generated by LinkML对此专门进行了解释。
2.4、Runtime
NMDC的这套系统由一系列组件构成,称作runtime(microbiomedata/nmdc-runtime: runtime system for NMDC data management and orchestration)。
- Dagster orchestration:
- dagit - a web UI to monitor and manage the running system.
- dagster-daemon - a service that triggers pipeline runs based on time or external state.
- PostgresSQL database - for storing run history, event logs, and scheduler state.
- workspace code
- Code to run is loaded into a Dagster
workspace. This code is loaded from one or more dagsterrepositories. Each Dagsterrepositorymay be run with a different Python virtual environment if need be, and may be loaded from a local Python file orpip installed from an external source. In our case, each Dagsterrepositoryis simply loaded from a Python file local to the nmdc-runtime GitHub repository, and all code is run in the same Python environment. - A Dagster repository consists of
solidsandpipelines, and optionallyschedulesandsensors.solidsrepresent individual units of computationpipelinesare built up from solidsschedulestrigger recurring pipeline runs based on timesensorstrigger pipeline runs based on external state
- Each
pipelinecan declare dependencies on any runtimeresourcesor additional configuration. There are TerminusDB and MongoDBresourcesdefined, as well aspresetconfiguration definitions for both “dev” and “prod”modes. Thepresets tell Dagster to look to a set of known environment variables to load resources configurations, depending on themode.
- Code to run is loaded into a Dagster
- A TerminusDB database supporting revision control of schema-validated data.
- A MongoDB database supporting write-once, high-throughput internal data storage by the nmdc-runtime FastAPI instance.
- A FastAPI service to interface with the orchestrator and database, as a hub for data management and workflow automation.
2.5、Workflow
NMDC的这套系统还支持提交数据后自动触发workflow进行生信pipeline分析,这个系统叫NMDC EDGE1。NMDC EDGE是一个用开源的EDGE Bioinformatics1框架搭建的、运行在HPC上的、有WebUI的生物信息计算平台;
另外,NMDC的workflow使用WDL语言描述,我们也可以自己部署支持WDL的workflow引擎来执行,如Cromwell。

3、中国NMDC
中国的NMDC是中科院微生物所牵头,委托北京极道科技建设的数据协作平台,不仅包含元数据提交、在线分析工具,还提供了各种常见的公开数据库下载。不过,因为该平台并非开源,所以能看到的信息很少,但其底层所用的技术组件可以从这里得知一二。